
🙆♂️ 병렬 통신
NetCon으로 병렬 통신을 합니다. NetCon 소스들은 임계값 감지기 입니다. 막 전위 등 변수들을 모니터링하고 임계값에 도달하면 이벤트가 발동됩니다. like 시냅스?
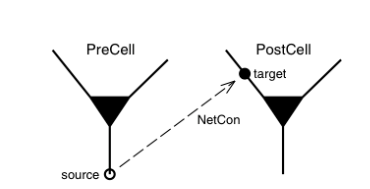
시냅스전 뉴런 "PreCell"에 부착된 "NetCon"은 소스라고 표시된 위치에서 스파이크를 감지하고 시냅스 후 뉴런 "PostCell"에 부착된 시냅스 "타겟"에 이벤트를 전달합니다.
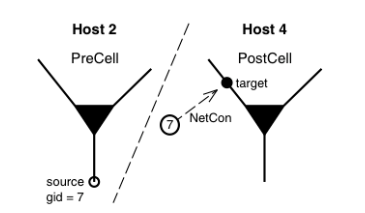
gid=7인 시냅스 전 스파이크 소스 "PreCell"은 "호스트 2"에 있지만 대상은 "호스트 4"의 "PostCell"에 연결된 시냅스입니다. "PreCell"이 급증하면 소스가 "gid 7"인 "NetCons"가 이벤트를 대상에 전달할 수 있도록 메시지가 모든 호스트에 전달됩니다.
🙋♂️ 실습 시작
MPI라는 도구를 사용할 것인데 Jupyter Notebook으로는 안됩니다.
https://neuron.yale.edu/ftp/neuron/2019umn/neuron-quickstart.pdf
위 문서 3슬라이드를 통해 MPI를 설치할 수 있습니다.
from neuron import h
h.nrnmpi_init() # initialize MPI
pc = h.ParallelContext()
print('I am {} of {}'.format(pc.id(), pc.nhost()))
h.quit() # necessary to avoid a warning message on parallel exit on some systems설치가 완료되었다면 위의 코드를 짜서 실험 코드를 실행해 볼 수 있습니다.
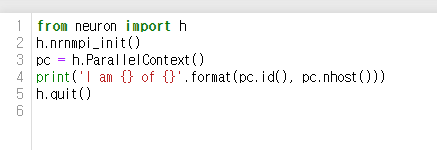
testmpi.py 파일을 만들어주고
터미널을 열고 해당 파일이 있는 경로로 이동하여
mpiexec -n 4 python testmpi.py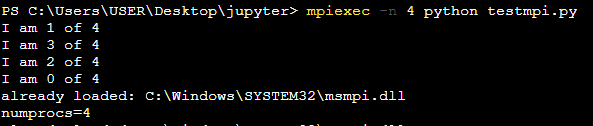
명령어를 쳐주면 4번 실행되는데 병렬 실행이라 순서대로 실행되는 것이 아닌 동시에 실행되는 모습입니다.
이제 실습을 위해 여러 파일을 만들 것입니다. 먼저 cell.py입니다.
from neuron import h
class Cell:
def __init__(self, gid, x, y, z, theta):
self._gid = gid
self._setup_morphology()
self.all = self.soma.wholetree()
self._setup_biophysics()
self.x = self.y = self.z = 0
h.define_shape()
self._rotate_z(theta)
self._set_position(x, y, z)
# everything below here in this method is NEW
self._spike_detector = h.NetCon(self.soma(0.5)._ref_v, None, sec=self.soma)
self.spike_times = h.Vector()
self._spike_detector.record(self.spike_times)
self._ncs = []
self.soma_v = h.Vector().record(self.soma(0.5)._ref_v)
def __repr__(self):
return '{}[{}]'.format(self.name, self._gid)
def _set_position(self, x, y, z):
for sec in self.all:
for i in range(sec.n3d()):
sec.pt3dchange(i,
x - self.x + sec.x3d(i),
y - self.y + sec.y3d(i),
z - self.z + sec.z3d(i),
sec.diam3d(i))
self.x, self.y, self.z = x, y, z
def _rotate_z(self, theta):
"""Rotate the cell about the Z axis."""
for sec in self.all:
for i in range(sec.n3d()):
x = sec.x3d(i)
y = sec.y3d(i)
c = h.cos(theta)
s = h.sin(theta)
xprime = x * c - y * s
yprime = x * s + y * c
sec.pt3dchange(i, xprime, yprime, sec.z3d(i), sec.diam3d(i))
다음은 ballandstick.py 입니다.
from neuron import h, gui
from neuron.units import ms, mV
from cell import Cell
class BallAndStick(Cell):
name = 'BallAndStick'
def _setup_morphology(self):
self.soma = h.Section(name='soma', cell=self)
self.dend = h.Section(name='dend', cell=self)
self.dend.connect(self.soma)
self.soma.L = self.soma.diam = 12.6157
self.dend.L = 200
self.dend.diam = 1
def _setup_biophysics(self):
for sec in self.all:
sec.Ra = 100 # Axial resistance in Ohm * cm
sec.cm = 1 # Membrane capacitance in micro Farads / cm^2
self.soma.insert('hh')
for seg in self.soma:
seg.hh.gnabar = 0.12 # Sodium conductance in S/cm2
seg.hh.gkbar = 0.036 # Potassium conductance in S/cm2
seg.hh.gl = 0.0003 # Leak conductance in S/cm2
seg.hh.el = -54.3 # Reversal potential in mV
# Insert passive current in the dendrite
self.dend.insert('pas')
for seg in self.dend:
seg.pas.g = 0.001 # Passive conductance in S/cm2
seg.pas.e = -65 # Leak reversal potential mV
# NEW: the synapse
self.syn = h.ExpSyn(self.dend(0.5))
self.syn.tau = 2 * ms
마지막으로 ring.py입니다.
from neuron import h
from ballandstick import BallAndStick
### MPI must be initialized before we create a ParallelContext object
h.nrnmpi_init()
pc = h.ParallelContext()
class Ring:
"""A network of *N* ball-and-stick cells where cell n makes an
excitatory synapse onto cell n + 1 and the last, Nth cell in the
network projects to the first cell.
"""
def __init__(self, N=5, stim_w=0.04, stim_t=9, stim_delay=1, syn_w=0.01, syn_delay=5, r=50):
"""
:param N: Number of cells.
:param stim_w: Weight of the stimulus
:param stim_t: time of the stimulus (in ms)
:param stim_delay: delay of the stimulus (in ms)
:param syn_w: Synaptic weight
:param syn_delay: Delay of the synapse
:param r: radius of the network
"""
self._N = N
self.set_gids() ### assign gids to processors
self._syn_w = syn_w
self._syn_delay = syn_delay
self._create_cells(r) ### changed to use self._N instead of passing in N
self._connect_cells()
### the 0th cell only exists on one process... that's the only one that gets a netstim
if pc.gid_exists(0):
self._netstim = h.NetStim()
self._netstim.number = 1
self._netstim.start = stim_t
self._nc = h.NetCon(self._netstim, pc.gid2cell(0).syn) ### grab cell with gid==0 wherever it exists
self._nc.delay = stim_delay
self._nc.weight[0] = stim_w
def set_gids(self):
"""Set the gidlist on this host."""
#### Round-robin counting.
#### Each host has an id from 0 to pc.nhost() - 1.
self.gidlist = list(range(pc.id(), self._N, pc.nhost()))
for gid in self.gidlist:
pc.set_gid2node(gid, pc.id())
def _create_cells(self, r):
self.cells = []
for i in self.gidlist: ### only create the cells that exist on this host
theta = i * 2 * h.PI / self._N
self.cells.append(BallAndStick(i, h.cos(theta) * r, h.sin(theta) * r, 0, theta))
### associate the cell with this host and gid
for cell in self.cells:
pc.cell(cell._gid, cell._spike_detector)
def _connect_cells(self):
### this method is different because we now must use ids instead of objects
for target in self.cells:
source_gid = (target._gid - 1 + self._N) % self._N
nc = pc.gid_connect(source_gid, target.syn)
nc.weight[0] = self._syn_w
nc.delay = self._syn_delay
target._ncs.append(nc)cell.py와 ballandsitck.py의 코드 내용은 전과 비슷합니다.
ring.py도 전에와 비슷하지만 ParalleCntext()를 사용하는 차이점이 있습니다.
첫 번째 테스트를 할 수 있습니다.
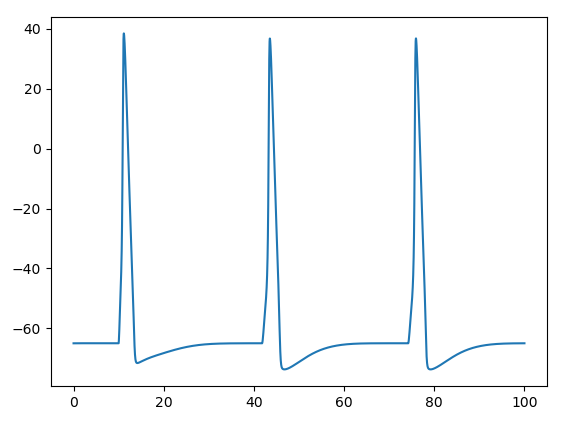
from neuron import h
from neuron.units import ms, mV
import matplotlib.pyplot as plt
from ring import Ring
cell_to_plot = 0
ring = Ring()
pc = h.ParallelContext()
pc.set_maxstep(10 * ms)
t = h.Vector().record(h._ref_t)
h.finitialize(-65 * mV)
pc.psolve(100 * ms)
if pc.gid_exists(cell_to_plot):
plt.figure()
plt.plot(t, pc.gid2cell(cell_to_plot).soma_v)
plt.show()
pc.barrier()
pc.done()
h.quit()test_ring1.py로 저장하고
python test_ring1.py실행할 수 있습니다.
'AI > neuron' 카테고리의 다른 글
| [neuron][파이썬] 22. BallAndStick 여러가지 실험 (0) | 2022.07.31 |
|---|---|
| [neuron][파이썬] 21. 깃헙 뉴런 튜토리얼 - Ball and Stick model (0) | 2022.07.27 |
| [neuron][파이썬] 21. 깃헙 뉴런 튜토리얼 - Single compartment neuron model (0) | 2022.07.27 |
| [neuron][파이썬] 18. 확장된 링 네트워크 (0) | 2022.07.25 |
| [neuron][파이썬] 17. 링 네트워크 시뮬레이션 실행 및 출력 (0) | 2022.07.22 |